
据EDN电子技术设计报道,《nature》发布了2024年值得关注的七大技术,分别为人工智能设计蛋白质、深度换脸检测、大片段DNA插入、脑机接口、超高分辨率显微成像、细胞图谱、3D打印纳米材料。

其中,入选的大片段DNA插入技术由北京中国科学院高彩霞领导的研究人员开发,这也是自2018年首次评选以来,第一项来自中国学者的技术成果入选。
2023 年末,美国和英国监管机构批准了首个基于 CRISPR 的基因编辑疗法,用于治疗镰状细胞病和输血依赖性 β 地中海贫血,这是基因组编辑作为临床工具的重大胜利。
CRISPR 及其衍生物使用短的可编程 RNA 将 DNA 切割酶(例如 Cas9)引导至特定的基因组位点。它们通常在实验室中用于禁用有缺陷的基因并引入小的序列变化。精确且可编程地插入跨越数千个核苷酸的较大 DNA 序列是很困难的,但新兴的解决方案可以让科学家替换有缺陷基因的关键片段或插入功能齐全的基因序列。
对于人体细胞的体内修饰,SSAP 可能提供更紧凑的解决方案:体积更大的 PASTE 机器需要三个独立的病毒载体进行传递,这可能会降低相对于双组件 SSAP 系统的编辑效率。也就是说,即使相对低效的基因替代策略也足以减轻许多遗传疾病的影响。
这些方法不仅与人类健康相关。
2023年4月,中国科学院遗传与发育生物学研究所高彩霞团队在 Nature Biotechnolgy 期刊发表了题为:Precise integration of large DNA sequences in plant genomes using PrimeRoot editors 的研究论文。
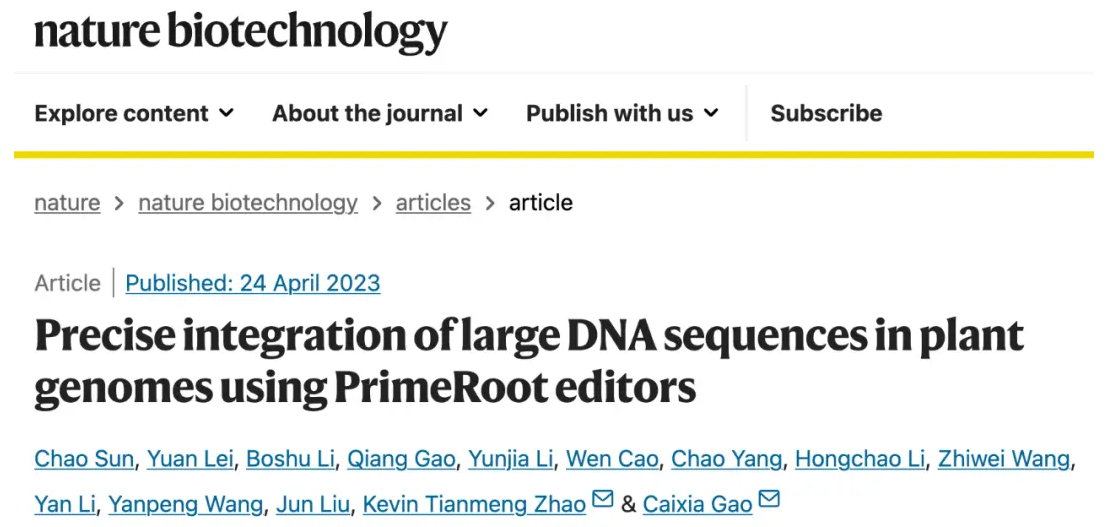
该研究将团队之前开发的ePPE(Engineered Plant Prime Editor)与刘如谦团队开发的epegRNA(Engineered pegRNA)结合,在植物细胞内建立了dual-ePPE系统,实现了最高效率可达50%以上的短片段DNA的精准定点插入。然后将dual-ePPE与筛选出的高效的酪氨酸家族位点特异性重组酶Cre相结合,开发了能够实现大片段DNA精准插入的PrimeRoot系统。该系统在水稻和玉米中能够实现一步法大片段DNA的精准定点插入,效率可达6%,成功插入的片段长度最长达11.1kb,且插入完全精准可预测,在编辑效率和精准性上具有显著优势。

高彩霞研究员表示,PrimeRoot系统高效、精准插入大片段DNA的能力,可通过基因敲入广泛用于赋予作物对疾病和病原体的抗性,从而继续推动基于CRISPR的植物基因组工程的创新浪潮。相信这项新技术可以应用于任何植物物种。
20年前,华盛顿大学的 David Baker 和他的同事实现了一项里程碑式的壮举::他们使用计算工具从头设计了一个全新蛋白质——“Top7”,该蛋白由93个氨基酸残疾组成,能够如预期般折叠,且非常稳定,但它没有任何有意义的生物学功能。
而现在,在 David Baker 等人的努力下,从头设计蛋白质已经一种成熟的工具,用于生成定制酶,及其他基于蛋白质的药物、疫苗和药物递送载体。这种进步的大部分归因于越来越多的将蛋白质序列与其结构联系起来的数据库,但人工智能的技术进步也同样重要。
公开可用的生成人工智能算法的爆炸式增长使得合成令人信服但完全人工的图像、音频和视频变得简单,结果出现了许多人工智能生成的与军事冲突有关的“Deepfake”图像和音频。
纽约布法罗大学的计算机科学家 Siwei Lyu 表示,他看到了大量由人工智能生成的与以色列-哈马斯冲突相关的“深度伪造”图像和音频。这只是一场高风险的猫鼠游戏的最新一轮,人工智能用户制作欺骗性内容,Lyu 和其他媒体取证专家致力于检测和拦截它。
一种解决方案是,生成式人工智能开发人员在模型输出中嵌入隐藏信号,为人工智能生成的内容打上水印。其他策略则侧重于内容本身。例如,一些经过处理的视频将某个公众人物的面部特征替换成了另一个人的面部特征,而新算法可以识别出被替换特征边界上的人工痕迹,Lyu 说。一个人外耳的独特褶皱也能揭示脸部和头部的不匹配,而牙齿的不规则则能揭示经过剪辑的唇语视频,在这些视频中,一个人的嘴部经过数字处理,说出了一些当事人没有说过的话。人工智能生成的照片也是一个棘手的挑战,也是一个不断变化的目标。
2023年8月,Francis Willett 团队以:A high-performance speech neuroprosthesis 为题,在Nature 期刊上发表了关于使用脑机接口让渐冻症患者发声的临床试验。

该论文显示,通过植入皮质内脑机接口(iBCI),并通过训练人工智能(AI)软件,能够将渐冻症(ALS)患者 Pat Bennett 大脑中的神经活动实时转化为文字,转化速度可达每分钟62个单词,总词汇量高达125000,相比已有的脑机接口速度更快、准确性更高、词汇覆盖率更大。 这项研究展示了一条可行的路径以恢复渐冻症等瘫痪者的语言沟通能力。
Stefan Hell、Eric Betzig 和 William Moerner 因打破限制光学显微镜空间分辨率的“衍射极限”而荣获 2014 年诺贝尔化学奖,由此得以在数十纳米级分辨率下进行分子尺度的成像实验。尽管如此,一些研究人员仍然渴望更好的结果——而且他们正在迅速取得进展。
2023年,德国普拉内格马克斯·普朗克生物化学研究所的纳米技术研究员 Ralf Jungmann 团队开发了一种序列成像(RESI)的增强分辨率的方法,可以分辨出DNA链上的单个碱基对,使用标准的荧光显微镜实现了埃级分辨率。
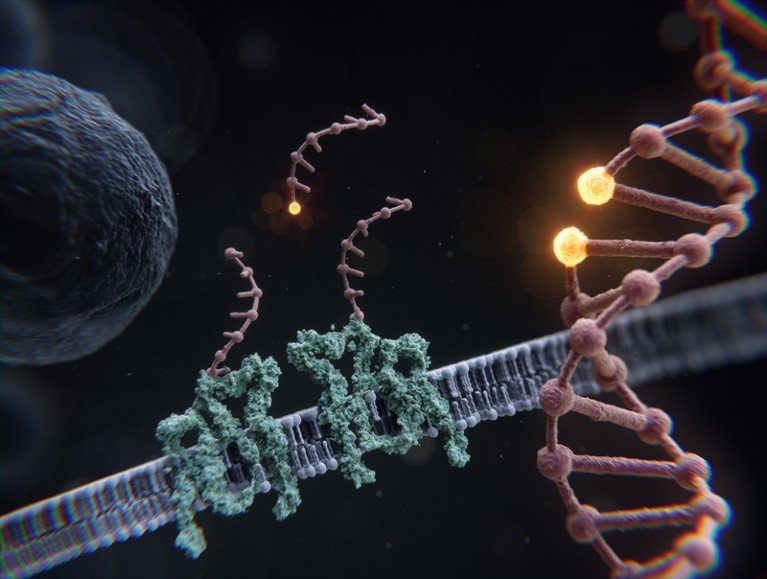
其中单个分子被不同的 DNA 链标记。然后用染料标记的互补 DNA 链检测这些分子,这些 DNA 链短暂但重复地与其相应的目标结合,从而可以区分单个荧光“闪烁”点,如果同时成像,这些“闪烁”点会模糊成单个斑点。这种通过顺序成像 (RESI) 方法增强分辨率的方法可以解析 DNA 链上的单个碱基对,从而用标准荧光显微镜展示埃级分辨率。
英国欣克斯顿 Wellcome Sanger 研究所的细胞生物学家 Sarah Teichmann 和加利福尼亚州南旧金山生物技术公司 Genentech 现任研究和早期开发负责人 Aviv Regev 于 2016 年发起一项规模庞大、雄心勃勃的人类细胞图谱 (Human Cell Atlas,HCA) 计划。该计划有近100个国家的约3000名科学家参与,而HCA本身也是一个更广泛的细胞和分子图谱交叉生态系统的一部分,包括人类生物分子图谱计划(HuBMAP)和脑计划(BICCN)。
去年,已有数十项研究展示了使用这些技术生成器官特异性图谱的进展。例如,6 月份,HCA 发布了对人肺 49 个数据集的综合分析。
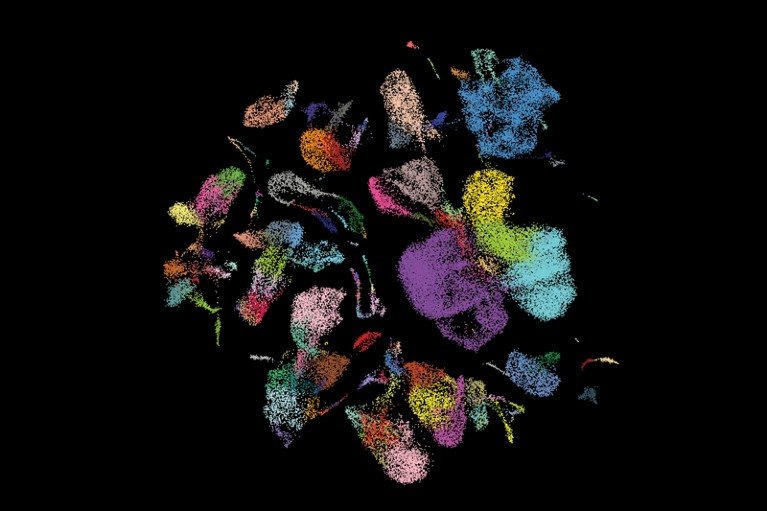
2023年,Nature 发布了一个论文集 (go.nature.com/3vbznk7) ,重点介绍了HuBMAP的进展,Science 则发布了一篇论文集,详细介绍了BICCN的工作 (go.nature.com/3nsf4ys) 。
Sarah Teichmann 表示,还有相当多的工作要做,估计至少需要五年时间才能完成HCA计划。但当该计划完成时,产生的人类细胞图谱将是无价之宝。例如可以使用细胞图谱数据来指导组织和细胞特异性药物开发,还有助于了解癌症等复杂疾病的风险和病因。
在纳米尺度上可能会发生奇怪而有趣的事情。这可能会使材料科学预测变得困难,但这也意味着纳米级建筑师可以制造具有独特特性的轻质材料,例如增加强度、与光或声音的定制相互作用以及增强的催化或能量存储能力。
存在多种精确制造此类纳米材料的策略,其中大多数使用激光来诱导光敏材料的图案化“光聚合”,并且在过去几年中,科学家在克服阻碍更广泛采用这些方法的限制方面取得了相当大的进展。
目前3D打印纳米材料还存在几个挑战,首先是速度, 使用光聚合技术组装纳米结构的速度大约比其他纳米级3D打印方法快三个数量级。这一速度可能已经足够用于实验室使用,但对于大规模生产或工业流程来说还是太慢了。
第二个挑战是并非所有材料都可以直接通过光聚合来打印,例如金属。但2022年,加州理工学院的 Julia Greer 开发出一种替代方法,将光聚合水凝胶作为微尺度模板,然后注入金属盐并进行处理,使金属在收缩的同时具有模板的结构。

最后是成本,这可能是最难突破的挑战,许多光聚合方法中使用的脉冲激光系统成本超过50万美元,但好在更便宜的替代品正在出现。
 最前沿的电子设计资讯
最前沿的电子设计资讯